打开黑匣——MRI数据是什么样的?
MRI数据是磁共振设备为大脑拍摄的“照片”,直观的理解它就是很多幅图片。
比如一张结构像:
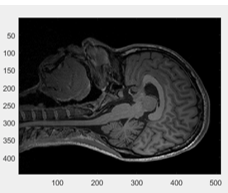
它是一张灰度图片,反映在数据上是这样的
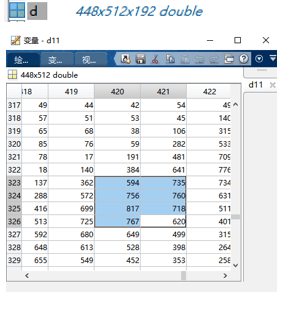
很多不同角度的图片叠在一起,就形成了一个3d“无死角”的大脑结构。
用MATLAB来认识MRI数据非常合适,MATLAB内置dicom库,可以直接调用。它可以读取dcm、IMA等格式的图像。
1 | filename = 'a.dcm'; |
dicomread 读取数据,然后用imagesc(img)即可可视化。
对于静息态来说,一张dcm读取进来的数据是二维的,但包含的却是三维的信息。而对于结构像T1或者DTI,则是二维的一个截面图像。
dicominfo包含该影像的所有信息,比如做任务态或者静息态的sliceorder信息
例如hearder.Private_0019_1029中就包含了每个slice的扫描时刻,这个信息可以用来判断sliceorder或者做多通道的slicetiming。
而转换过后的nii格式的数据,则是3维或者4维的。
nii格式的可以通过spm的命令读取
1 | % read Nii data to matlab workspace |
既然是3D的数据,我们可以从各个角度观察它
1 | [x,y,z]=meshgrid(1:512,1:448,1:192); |

打开黑匣——MRI数据是什么样的?